Introduction
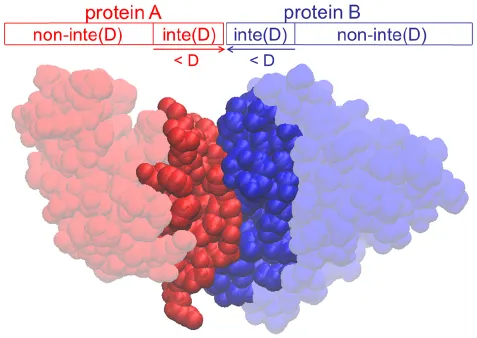
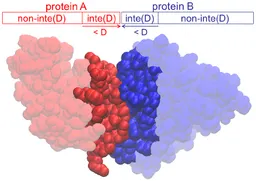
단백질 구조를 다루다 보면, 두 개 이상의 단백질끼리 결합하거나, 가까운 거리에서 상호작용하는 경우가 있다. 이때 단백질끼리 인접한 부분의 residue를 interface 라고 부른다.
이번 포스팅에서는 크게 두 가지 함수를 소개한다.
1.
get_interface(): Interface 부분을 얻는 함수
2.
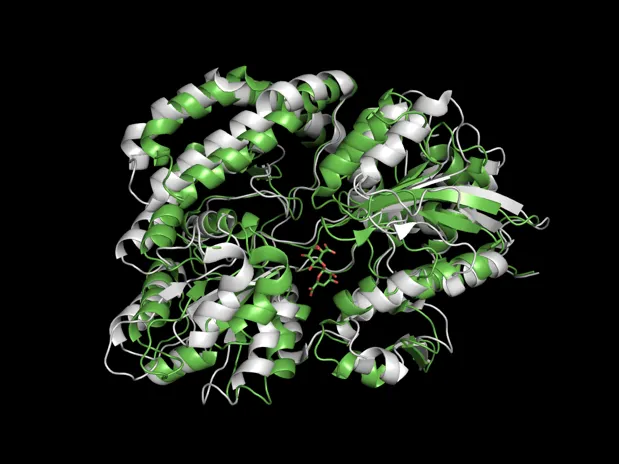
get_interface_rmsd(): Ground truth bound structure의 interface 부분과 rigid docking model이 예측한 bound structure의 interface 부분 간 RMSD 값을 얻는 함수
Implementation with Python
import scipy.spatial as spa
import torch
def get_interface(A, B, threshold=8.):
"""
@params
- A, B: coordinates (torch.tensor, shape: (N x 3))
- threshold: threshold distance to define interface (float). usually 8 angstrom.
"""
a_b_distance = spa.distance.cdist(A, B)
positive_tuple = np.where(a_b_distance < threshold)
return positive_tuple
Python
복사
def get_interface_rmsd(lig_coords_pred, lig_coords_true, rec_coords, threshold):
"""
@params
- lig_coords_pred, lig_coords_true,
"""
lig_inter, rec_inter = get_interface(lig_coords_true, rec_coords, threshold)
lig_coords_pred = lig_coords_pred[lig_inter, :]
lig_coords_true = lig_coords_true[lig_inter, :]
rec_coords = rec_coords[rec_inter, :]
complex_coords_pred = torch.concatenate([lig_coords_pred, rec_coords], dim=0)
complex_coords_true = torch.concatenate([lig_coords_true, rec_coords], dim=0)
R, t = Kabsch_align(complex_coords_pred, complex_coords_true)
complex_coords_pred_aligned = (R @ complex_coords_pred.T + t).T
interface_rmsd = compute_rmsd(complex_coords_pred_aligned, complex_coords_true)
return interface_rmsd
Python
복사
•